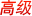Illumina测序原理—NGS技术
原理上,illumina NGS技术类似桑格测序。DNA合成测序循环中,DNA聚合酶催化荧光标记的dNTPs到DNA模板链上。每次循环,dNTP被掺入的同时,核苷被荧光激发识别。但zui关键的不同是,不同于合成完后测序整个DNA片段,NGS通过平行测序模式将过程高通量化了。
Illumina NGS流程包括四个步骤:
1. 文库制备
DNA或cDNA样本随机片段化后,5′端和 3′端连接接头。连接好接头的DNA片段进行PCR扩增和纯化。

图A 文库制备。基因组DNA—片段化—接头连接—得到测序文库
NGS 文库制备:片段化基因组DNA,然后在DNA片段两端连接特殊接头。
2. 簇生成
文库上样到流动槽,DNA片段通过接头和槽内寡核苷酸配对连接。接着,每个片段通过桥式法扩增成簇克隆。之后,就可以测序了。

图B:簇扩增
3. 测序
Illumina SBS 技术(边合成边测序)在单个碱基被合成到DNA模板链的同时识别它。因为每个测序循环都整合4种dNTPs,自然竞争机制使得偏差zui小化,相比其他测序技术原始错误率低很多。即使用于序列重复区域和同聚物,碱基测序准确率也是很高的。

图C 测序 测序试剂,包括荧光标记的核苷酸,加入后合成。流动槽有图像功能的,每个簇的荧光激发都会被记录。激发的波长和强度可以识别碱基。这一循环重复N次,得到读长为N个碱基的序列。
4. 数据分析
数据分析和比对过程中,测序序列(reads)与参考基因组进行比对,能检测到多种基因突变,比如单核苷酸多态性(SNP)或DNA插入缺失(indel),RNA,系统发育(phylogenetic)或 宏基因组学分析等。

图D:序列比对和数据分析 序列(Reads)通过生物信息学软件和参考基因组比对。比对后,就能识别新测序的序列(reads)和参考基因组的不同。
Illumina*SBS技术视频
http://v.youku.com/v_show/id_XMTI1MjA5Mzg5Mg==.html?spm=a2hzp.8253869.0.0
原文 illumina*资料
Illumina NGS workflows include four basic steps:
1. Library Preparation—The sequencing library is prepared by random fragmentation of the DNA or cDNA sample, followed by5′and 3′adapterligation (Figure 3A). Alternatively,“tagmentation” combines the fragmentation and ligation reactions into a single step that greatly increases the efficiency of the library preparation process.9 Adapter-ligated fragments are then PCR amplified and gelpurified.
2. Cluster Generation—For cluster generation, the library isloaded into a flow cell where fragments are captured on a lawn of surface-bound oligos complementary to the library adapters. Each fragment is then amplified into distinct, clonalclusters through bridge amplification (Figure 3B). When cluster generation is complete, the templates are ready for sequencing.
3 Sequencing—Illumina SBS technology uses a proprietary reversible terminator–based method that detects single bases as they are incorporated into DNA template strands(Figure 3C). As all four reversible terminator–bound dNTPs are present during each sequencing cycle, natural competition minimizes incorporation bias and greatly reduces raw error rates compared to other technologies.6,7 The result is highly accurate base-by-base sequencing that virtually eliminates sequence context–specific errors, even within repetitive sequence regions and homopolymers.
5. Data Analysis—During data analysis and alignment, the newly identified sequence reads are aligned to a reference genome (Figure 3D). Following alignment, many variations of analysis are possible, such as single nucleotide polymorphism (SNP)or insertion-deletion (indel) identification, read counting for RNA methods, phylogenetic or metagenomic analysis, and more.
基因测序背景知识-illumina版
DNA测序简介
Illumina下一代测序(NGS)技术使用克隆扩增和边合成边测序(sequencing by synthesis, SBS)的方法来实现快速、 准确的测序。这一过程在将DNA碱基加入核酸链的同时对其进行识别。每个碱基在加入不断延伸的链的同时发出*的荧光信号,这些信号可以被用来确定DNA序列的顺序。
NGS 技术可以用来对任何一种生物的DNA进行测序,提供有价值的信息,解答几乎任何的生物学问题。作为一种高度可扩展的技术,DNA 测序可以通过多种方法应用于小的、靶向的区域或整个基因组,有利于研究人员调查,并更好地了解健康和疾病。
通过NGS开展DNA测序的好处
以大规模并行的方式测序大段的DNA,与基于毛细管电泳的Sanger测序相比,具有通量和规模上的优势
提供高分辨率,可在到每个碱基的水平上查看基因、外显子组或基因组
根据信号强度可实现定量测定
可检测几乎所有类型的基因组改变,包括单核苷酸变异、插入和缺失、拷贝数变化和染色体畸变
为扩展研究和同时测序多个样品提供高通量和灵活性
主要的DNA测序方法
全基因组测序
全基因组测序是分析基因组的zui全面的方法。迅速降低的测序成本,以及从完整遗传密码中获得宝贵信息的能力,使得全基因组测序成为一种强大的研究工具。
靶向重测序
通过靶向重测序,一组基因或基因组区域被分离出来并测序,让研究人员能够将时间、经费和分析集中在感兴趣的特定区域上。
ChIP测序
通过将染色质免疫沉淀(ChIP)检测和测序相结合,ChIP测序(ChIP-Seq)是鉴定转录因子及其他蛋白在全基因组范围内的DNA结合位点的有力方法。
DNA测序主要应用
癌症DNA测序
利用NGS进行癌症DNA测序,可为全基因组研究、靶向的基因图谱分析、肿瘤-正常样本的比较等研究提供信息。NGS所提供的灵敏度,足以用于检测稀有体细胞变异、肿瘤亚克隆和循环DNA片段。

微生物测序
利用DNA测序来分析微生物物种,可为环境宏基因组学研究、传染病监测、分子流行病学等研究提供信息。

致病变异的发现
高通量的DNA测序技术可实现大规模数量样本的快速筛查,从而发现与复杂疾病相关的致病变异。

Illumina试剂产品信息欢迎您咨询 华雅再生医学旗舰公司:红荣微再(上海)生物工程技术有限公司